引言
在高浓度下,一些底物浓度也会抑制酶的活性。在所有已知酶中,约有 20% 会出现底物抑制现象。当两种底物分子能与酶结合,从而阻碍酶的活性时,就会出现这种情况。
步骤
创建 XY 数据表。在 X 列输入底物浓度,在 Y 列输入酶活性。如果有多个实验条件,则将第一个条件放入 A 列,第二个条件放入 B 列,等等。
输入数据后,单击分析,选择非线性回归,选择酶动力学方程面板,然后选择底物抑制。
模型
Y=Vmax*X/(Km + X*(1+X/Ki))
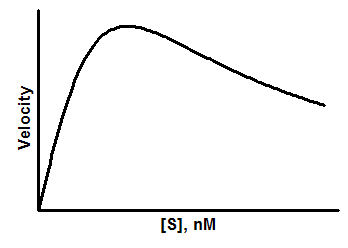
参数
Vmax是在底物不抑制酶活性的情况下的最大酶速,单位与 Y 相同。
Km 是迈克尔斯-门顿常数,单位与 X 相同。
Ki 是 底物结合的解离常数,即两种底物可以结合到一种酶上。其单位与 X 相同。
注意:Prism 7 及以后版本使用的初始值规则与 Prism 6 不同(经过改进)。
为什么该模型并不总是收敛拟合
如果将该模型重新排列,会更容易理解拟合问题:
Y=Vmax/(Km/X + 1 + X/Ki)
Vmax 控制着峰值的高度(但不等于峰值处的 Y 值)。
当 X 值较小时,最后一项趋近于零,因此曲线的早期部分(X 值较小)由 Km 值决定。
相反,当 X 值较大时,第一项趋近于零,因此曲线的后期部分(X 值较大)由 Ki 值决定。
曲线的中间部分则由 Ki 和 Km 共同决定。
有两个原因会导致拟合结果 "模糊"。
•没有足够大范围的 X 值数据点。需要有小于 Km 且大于 Ki 的 X 值,最好有很多 X 值。如果只有曲线中间的数据,就根本没有信息来分别确定 Km 和 Ki。
•曲线的形状虽然看起来像底物抑制曲线,但并不完全正确。换句话说,这些数据确实不符合底物抑制模型。