尽管非线性回归,顾名思义,是为拟合非线性模型而设计的,但有些推论实际上假定模型的某些方面接近线性,因此每个参数值的不确定性是对称的。
对方程重新参数化可以使不确定性更加对称,使 SE 更容易解读,也使对称渐近 CI 更有帮助。Prism 可以计算非对称 CI,当您选择这种方法时,如何对方程进行参数化就不那么重要了。
什么是重新参数化?
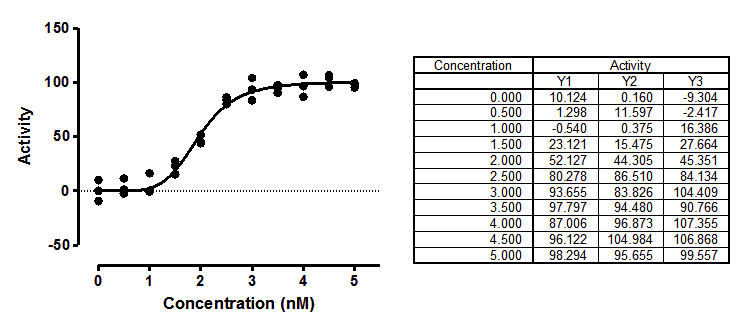
用于将曲线酶动力学数据拟合到标准模型的模型有两种形式:
Y=Vmax*X^h/(Khalf^h + X^h)
Y=Vmax*X^h/(Kprime + X^h)
这两个方程是等价的。它们都拟合 Vmax(外推到底物浓度非常高时的最大活性)和 h(Hill 斜率,描述曲线的陡峭程度)。但一个模型拟合的是 Khalf(获得最大速度一半所需的浓度),另一个模型拟合的是 Kprime(底物作用的一种更抽象的测量方法)。
哪个模型最好?两者是等价的,Kprime 等于Khalfh,因此它们会生成完全相同的曲线。
由于平方和将完全相同,自由度也将完全相同,因此,无论您选择这种模型的哪种形式,对这种模型和另一种模型进行任何比较都将得到完全相同的结果。
参数分布并不总是对称的
模拟可以确定参数不确定性的对称程度。我用 Vmax=100、h=5、Kprime=25(所以 Khalf=5)和 SD 等于 7.5 的高斯散射模拟了西格玛酶动力学。X 值与上图相符,每个 X 处的 Y 值为三份。我重复模拟了 5000 次,将每条曲线都拟合为两种形式的模型,并将 Kprime 和 Khalf 的拟合优度值列表,还计算了各自的偏斜度。
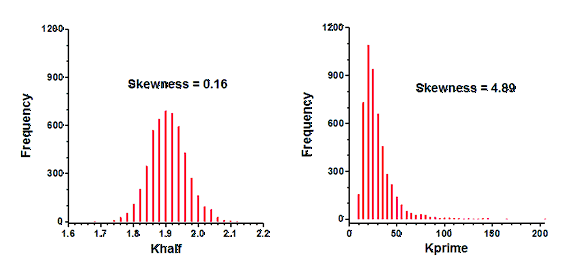
显然,Khalf 的分布相当对称,看起来是高斯分布。偏斜度接近零,符合对称分布的预期。相比之下,Kprime 的分布则相当偏斜度。请注意,有几次模拟的 Kprime 拟合优度值大于 100。偏斜度值(4.89)证实了通过观察可以明显看出的情况--分布远非对称。
用霍加尔偏斜度量化不对称现象
上述结果是通过多次模拟计算得出的。还有一种更简单的方法来计算参数的对称性。Prism 可以计算每个参数的霍加偏斜度,计算公式、数据点数量、X 值间距和参数值。对于模拟数据集,Khalf 的霍加偏斜度为 0.09,Kprime 的霍加偏斜度为 1.83。一个经验法则是,当霍加德偏斜度的绝对值大于 0.25 时,期望值会出现不对称问题;当该值大于 1.0 时,期望值会出现大问题。这些值可以通过一个没有模拟的数据集计算出来,它们告诉你,当你拟合 Khalf 时,对称置信区间会比拟合 Kprime 时更准确。
请注意,虽然 Prism 6 和 7 可以正确计算非加权拟合的 Hougaard 偏斜度,但如果您选择不等加权,它们的计算就会出错。Prism 8 修正了这一问题。
不对称参数的后果
理想情况下,置信区间易于解读。95% CI 有 95% 的概率包含参数的真实人口值,5% 的概率缺失参数值。
在分析真实数据时,我们永远不知道真实参数的值,因此永远不知道区间是否包含它。但模拟数据时,我们知道参数的真实值,因此可以量化置信区间的覆盖范围。我设置了上述相同的模拟,将每个数据集拟合到两个等式中,并将每个置信区间是否包含真实参数值列表。该表显示了在 5000 次模拟中,渐近对称置信区间不包括真实参数值的部分(Kprime 为 25,Khalf 为 1.9037)。
"95% CI |
"99% CI |
|
理想 |
5.0% |
1.0% |
Kprime |
8.8% |
4.8% |
Khalf |
5.1% |
1.0% |
这些结果表明,鉴于 Khalf 的对称性(见上文),Khalf 表现良好。95% 的置信区间期望值在 5.0% 的模拟中缺失真实值。事实上,只有 5.1% 的情况会出现这种情况。同样,期望值为 99% 的置信区间会在 1.0% 的模拟中缺失真实值,事实也正是如此。相比之下,Kprime 的表现就没那么好了。计算出的 95% 置信区间不够宽,因此在 8.8% 的模拟中缺失了真实值。99% 置信区间同样不够宽,因此有 4.8% 的模拟结果缺失了真实值。因此,计算得出的置信区间为 99%,实际结果为 95%。
这些模拟显示了选择适合 Khalf 的方程而不是适合 Kprime 的方程的优势。Khalf 的分布是对称的,因此根据这些拟合值计算出的置信区间可以按表面值来解读。相比之下,Kprime 的分布不对称,其置信区间不能从表面价值来解读。
如果要求 Prism 呈现非对称剖面似然置信区间,参数化并不重要
如果选择不对称剖面似然置信区间,那么选择哪种形式的方程并不重要。两种情况下的覆盖率是一样的,都非常接近 95% 或 99%。有了这个选择,你就可以选择符合教科书和论文的方程形式,或者符合你的思维方式的方程形式。如果你喜欢图形思维,请选择 Khalf。如果你喜欢机械地思考,请选择 Kprime。