引言
最常见的酶动力学实验是改变底物浓度并测量酶的速度。目的是确定酶的 Km(产生半最大速度的底物浓度)和 Vmax(最大速度)。如果您的目标是确定周转次数 kcat,而不是 Vmax,请使用该方程的另一个版本。
步骤
创建 XY 数据表。在 X 列输入底物浓度,在 Y 列输入酶的速度。如果您有多个实验条件,可将第一个条件放入 A 列,第二个条件放入 B 列,等等。
您也可以选择 Prism 的样本数据:酶动力学--迈克尔斯-门顿(Michaelis-Menten)。
输入数据后,点击分析,选择非线性回归,选择酶动力学方程面板,然后选择Michaelis-Menten 酶动力学。
模型
Y = Vmax*X/(Km + X)
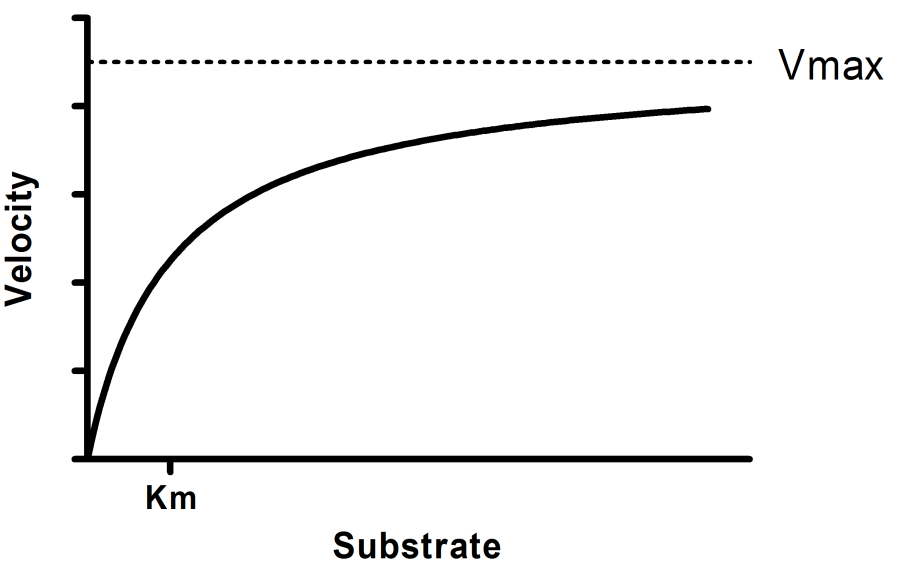
解读参数
Vmax是酶的最大速度,单位与 Y 相同。它是推断出底物浓度非常高时酶的速度,因此其值几乎总是高于实验中测得的任何速度。
Km是 Michaelis-Menten 常数,单位与 X 相同,是达到半最大酶速所需的底物浓度。
创建线性回归图
在非线性回归出现之前,研究人员必须将曲线数据转化为直线,以便使用线性回归进行分析。其中一种方法是绘制 Lineweaver-Burk 图,即底物浓度的倒数与酶速度的倒数对比图。
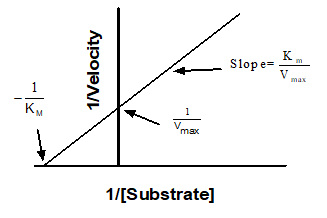
如果绘制 Lineweaver-Burk 图,只能用来显示数据。不要使用线性回归线的斜率和截距来确定 Vmax 和 Km 值。如果这样做,就无法获得最准确的 Vmax 和 Km 值。问题在于转换(倒数)会扭曲实验误差,因此双倒数图不符合线性回归的假设。使用非线性回归可获得最准确的 Km 和 Vmax 值。
要绘制 Lineweaver-Burk 图(带相应的直线),首先要记下非线性回归报告的 Vmax 和 Km 值(我们稍后会用到)。首先,从包含数据的数据表中单击工具栏上的 "分析"按钮,从 "变换、规范分析...... "分析部分选择 "变换",然后单击 "确定"。在 "功能列表"下拉菜单中选择 "药理学和生物化学转换"。选择 "Lineweaver-Burk"选项,然后点击确定(请确保已选中 "创建新的结果图"复选框)。
现在,我们要在 Lineweaver-Burk 图表中添加适当的线。切勿简单地执行线性回归,因为这不会生成上述的正确直线。请按照以下步骤操作:
1.在转换后的数据图表中,单击工具栏 "分析"部分的 "分析"按钮
2.从分析的 "生成曲线"部分选择 "绘制函数",然后单击确定
3.在出现的对话框中,从 "函数"选项卡的函数列表中选择 "直线
4.使用 "函数"选项卡底部的 "X 值范围"选项指定直线的起点和终点
5.切换到 "参数值和列标题"选项卡
6.计算 1/Vmax,并将此值作为 YIntercept 输入(其中 Vmax 是之前非线性回归报告的值)
7.输入 Km/Vmax 作为斜率(其中 Km 和 Vmax 是之前非线性回归报告的值)
8.点击确定
9.Prism 将生成一个名为 "绘制函数"的新数据表。单击并将该表拖动到 Lineweaver-Burk 图形上,然后单击 "添加"。
注释
•请参阅酶动力学所有分析的假设列表。
•该方程拟合的曲线与拟合周转数 Kcat而非 Vmax 的方程完全相同。 Kcat 乘以 Et(酶位点浓度)的乘积等于 Vmax,因此如果知道 Et,Prism 就能拟合 kcat。
•
•请注意,Km 并不是一个衡量酶与底物之间结合强度的结合常数。它的值考虑了底物对酶的亲和力,以及与酶结合的底物转化为产物的速率。