引言
Kcat 是周转数,即每个酶位点在单位时间内将底物分子转化为产物的数量。如果知道酶位点的浓度,在分析底物与速度的关系曲线时,就可以拟合 Kcat 而不是 Vmax。
模型
Y = Et*kcat*X/(Km + X)
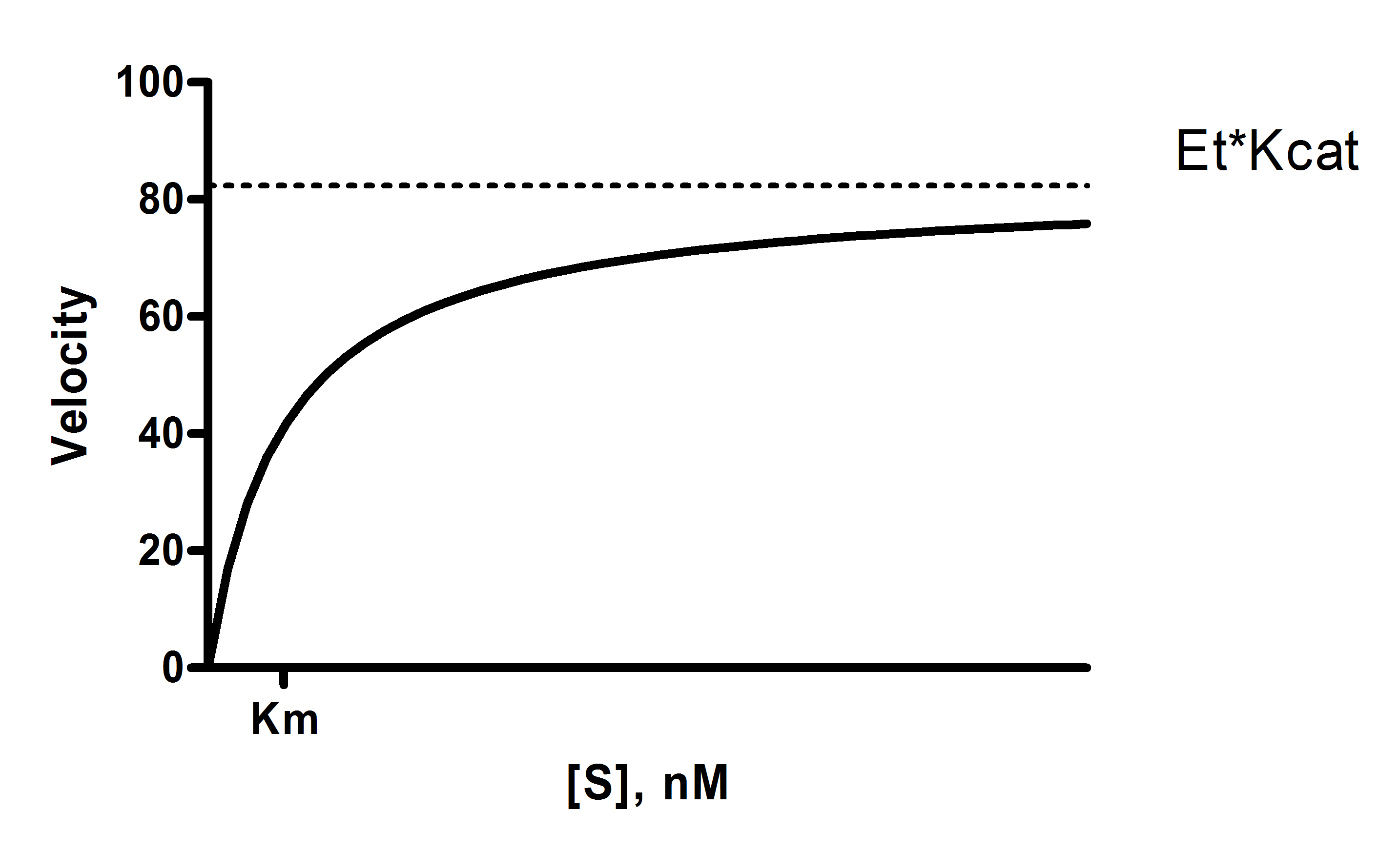
X 是底物浓度。
Y 是酶的速度。
kcat是周转次数,即每个酶位点在单位时间内将底物转化为产物的次数。它以 Y 轴时间单位的倒数表示。本示例中,如果 Y 的单位是微摩尔/分钟,那么 kcat 就是每个催化位点每分钟产生的底物分子数。
Km是迈克尔斯-门顿常数,单位与 X 相同。它是达到半最大酶促速度所需的底物浓度。
Et是酶催化位点的浓度。如果酶有多个亚基,请注意 Et 是催化位点的浓度,可能大于酶分子的浓度。您输入的 Y 值是以每次浓度为单位输入的酶速度。Et 必须以相同的浓度单位输入(而时间单位由 kcat 定义)。
Vmax是酶的最大速度,单位与 Y 相同。 它是推断底物浓度非常高时酶的速度,因此几乎总是高于实验中测得的任何速度。计算方法是 Et 乘以 kcat。
与迈克尔斯-门顿模型的关系
上图所示曲线与 Michaelis-Menten 模型定义的曲线完全相同。将任一模型拟合到数据中,都会得到完全相同的曲线,Km 值也完全相同。
Michaelis-Menten 模型可以找到 Vmax,即推断出底物浓度非常高时的最大酶速。它与您输入 Y 值(酶活性)时使用的单位相同。通常可以直接表示为(或转换为)摩尔/分钟/毫克蛋白质。Vmax 由存在多少酶位点(Et)和酶将底物转化为产物的速率(kcat)决定。
如果您知道添加到测定中的酶位点浓度(Et),那么您就可以使用上述模型拟合催化常数 Kcat。
在计算 Kcat 时,浓度单位会被抵消,因此 Kcat 是以逆时间单位表示的。它是周转数--每个酶位点在单位时间内将底物分子转化为产物的数量。
用 Prism 拟合模型
1.创建 XY 数据表。在 X 列输入底物浓度,在 Y 列输入酶的速度。如果有多个实验条件,则将第一个条件放入 A 列,第二个条件放入 B 列,等等。您也可以选择 Prism 的样本数据:酶动力学--迈克尔斯-门顿(Michaelis-Menten)。
2.输入数据后,点击分析,选择非线性回归,选择酶动力学方程面板,然后选择Kcat。
3.您必须根据其他实验将Et约束为一个常数。要限制 Et 值,请转到非线性回归对话框的 "限制 "选项卡,确保 Et 旁边的下拉菜单设置为 "等于常数",然后输入该值。对于样本数据,输入 100 作为 Et 值。
如果不知道 Et 值,就不能拟合 kcat,而应该拟合 Vmax。Prism 不可能同时拟合 kcat 和 Et 值,因为这两个参数是相互交织的,而底物-速度曲线没有给出它们各自的值。
4.根据样本数据,Prism 报告 Km= 5.886,95% 置信区间为 3.933 至 7.839。kcat 的拟合优度值为 13.53,95% 置信区间为 11.97 至 15.09。
注释
•参见所有酶动力学分析的假设列表。
•该方程拟合的曲线与拟合 Vmax 而非周转数 Kcat 的方程完全相同。
•