引言
如果酶具有合作亚基,则酶的速度与底物浓度的函数关系图将呈现半正态分布。Prism 提供了一个拟合底物-速度曲线的经验公式。请阅读有关酶动力学的高级书籍,了解基于别构作用分子模型的其他方法。
如何输入数据
创建 XY 数据表。在 X 列中输入底物浓度,在 Y 列中输入酶的速度。如果有多个实验条件,则将第一个条件放入 A 列,第二个条件放入 B 列,等等。
输入数据后,点击分析,选择非线性回归,选择酶动力学方程面板,然后选择变构 S 形酶动力学。
模型
Y=Vmax*X^h/(Khalf^h + X^h)
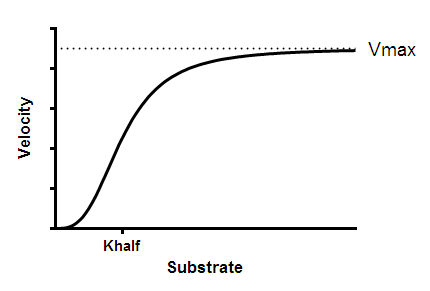
解读参数
Vmax是酶的最大速度,单位与 Y 相同。它是推断出底物浓度非常高时酶的速度,因此几乎总是高于实验中测得的任何速度。
Khalf是产生半最大酶速的底物浓度。它就是 EC50。
h是 Hill 斜率。当 h=1 时,该方程与标准Michaelis-Menten 方程相同。当 h 大于 1.0 时,由于正向协同作用,曲线呈曲线形。变异性 h 并不总是等于相互作用结合位点的数量,但其值不能超过相互作用位点的数量。可以把 h 看作是曲线陡峭程度和合作性存在与否的经验度量。
Kprime与 Km 有关。其计算公式为 Khalf^h,单位与 X 相同。
方程的另一种形式
该方程的另一个版本(下文引用的科普兰博士著作中的方程 5.47)与 Kprime 相符。
Y=Vmax*X^h/(Kprime + X^h)
请注意,该方程中的 Kprime 等于 Prism 内置方程中的 Khalf^h。这两个模型生成的曲线完全相同,只是报告参数的方法不同而已。Prism 同时报告 Kprime 和 Khalf。