什么是列常数?
当您同时拟合多个数据集时,可以将列标题作为第二个自变量。我们将此称为限制参数为列常数。本示例最能说明这一点。
如何输入列常数
要了解列常数的工作原理,请使用示例数据文件创建一个新的 XY 表:酶动力学 - 竞争性抑制。
数据表有一个 X 列和四个 Y 列,每个列代表不同浓度的抑制剂。抑制剂浓度作为列标题输入。
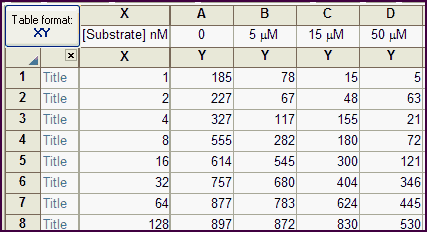
请注意,Prism 只读取列标题中的数字。在本示例中,单位指定为微摩尔,但 Prism 忽略了这一点,只读取数字。它不会进行任何单位转换。
拟合数据时指定列常数
选择初始值时,请从下拉列表中选择 "列标题平均值"或 "log(列标题平均值)"。
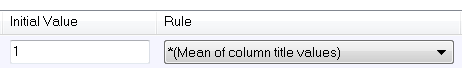
本示例
要拟合上述样本数据,请单击 "分析"(Analyze),选择非线性回归(Nonlinear regression),选择 "酶动力学"(Enzyme Kinetics)方程面板,然后选择 "竞争酶动力学"(Competitive enzyme kinetics)。方程已内置,但如果单击 "详细信息"按钮,就可以看到计算结果。
KmObs=Km(1+[I]/Ki)
Y=Vmax*X/(KmObs+X)
第一行定义了一个中间变异性变量(KmObs,存在竞争性抑制剂时观察到的迈克尔斯-门顿常数),它是酶的迈克尔斯-门顿常数(Km)、抑制剂浓度(I)和竞争性抑制常数(Ki)的函数。
第二行计算的酶速度(Y)是底物浓度(X)和 KMapp 的函数。
在定义该模型时,I 被限制为数据集常数,即其值来自列标题。因此,在本示例中,拟合 A 栏时 I=0,拟合 B 栏时 I=5,等等。Prism 会忽略标题中的 "μΜ"--它不会进行任何单位转换。
其他三个参数(Km、Ki 和 Vmax)被定义为共享参数,因此 Prism 拟合一个适用于整个数据集系列的拟合优度值。

Prism 确定了无抑制剂时酶的最大速度(Vmax,单位与您输入的 Y 值相同)、无抑制剂时酶的 Michaelis-Menten 常数(Km,单位与 X 值相同)和竞争性抑制常数(Ki,单位与 X 列常数相同)。请注意,I 并非拟合参数,而是采用您在列标题中输入的常数值。KmObs 不是拟合参数,而是用于定义模型的中间变异性变量。
了解有关竞争性酶抑制的更多信息。
总结。柱常数的优势
通过使用列常量和全局拟合(共享参数),本示例确定了一个参数(Ki),该参数的值无法通过任何一个数据集确定,只能通过研究数据集之间的关系来确定。